決定木
データ全体を説明変数を用いて段階的にグループ分けする手法。
決定木を書く
テキストではmvpartパッケージを使用しているが、現在は公開が停止されているので代わりにpartykitパッケージを使用する。
rpart()の代わりに使う関数はctree()であるが、引数の入れ方はほとんど変わらない。結果はpartyオブジェクトになっているので、そのままプロットすれば決定木が表示される。
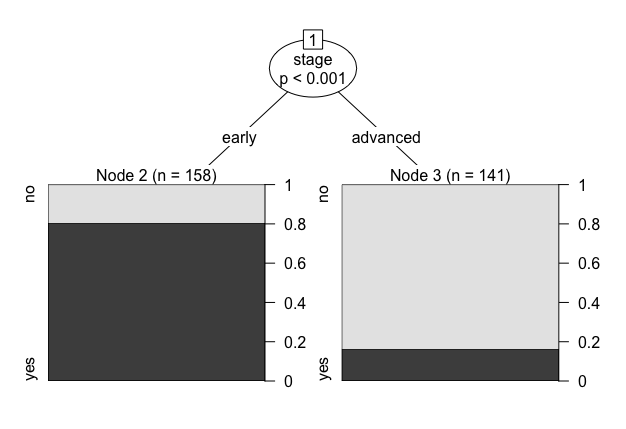
library(partykit) library(vcd) result <- ctree(survival ~ stage + operation + xray, weight = Freq, data = OvaryCancer) result # Model formula: # survival ~ stage + operation + xray # # Fitted party: # [1] root # | [2] stage in early: yes (n = 158, err = 19.6%) # | [3] stage in advanced: no (n = 141, err = 16.3%) # # Number of inner nodes: 1 # Number of terminal nodes: 2 plot(result)
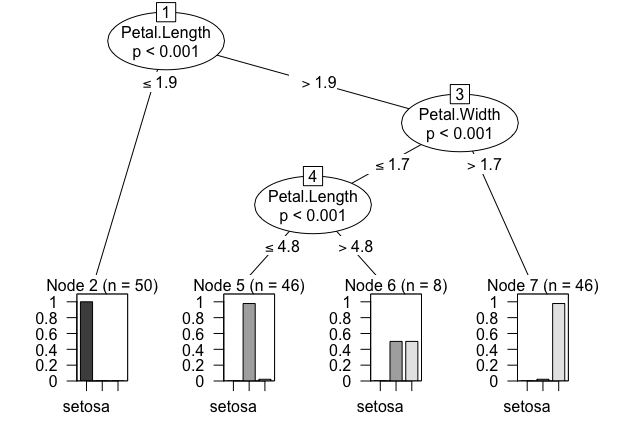
量的変数を用いる
ctree()を使った結果はテキストと若干異なる。
# 量的変数を用いたグループ分け data(iris) result <- ctree(Species ~ ., data = iris) result # Model formula: # Species ~ Sepal.Length + Sepal.Width + Petal.Length + Petal.Width # # Fitted party: # [1] root # | [2] Petal.Length <= 1.9: setosa (n = 50, err = 0.0%) # | [3] Petal.Length > 1.9 # | | [4] Petal.Width <= 1.7 # | | | [5] Petal.Length <= 4.8: versicolor (n = 46, err = 2.2%) # | | | [6] Petal.Length > 4.8: versicolor (n = 8, err = 50.0%) # | | [7] Petal.Width > 1.7: virginica (n = 46, err = 2.2%) # # Number of inner nodes: 3 # Number of terminal nodes: 4 plot(result)
また、predict()にtypeオプションの指定は必要ない。
iris$est <- predict(result) xtabs( ~ Species + est, data = iris) # est # Species setosa versicolor virginica # setosa 50 0 0 # versicolor 0 49 1 # virginica 0 5 45
順序カテゴリカルデータ
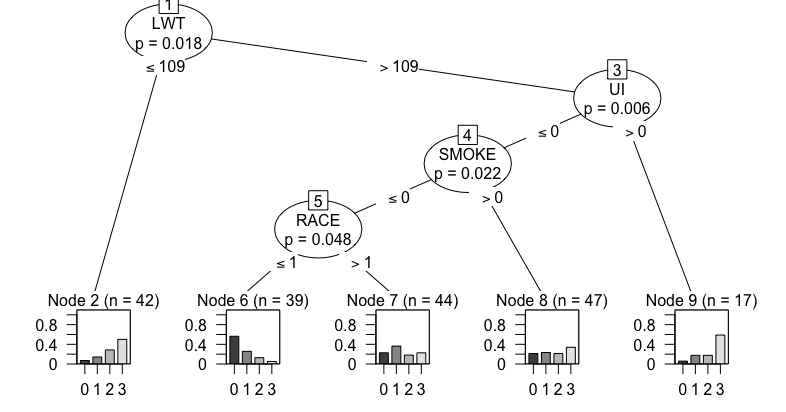
パッケージrpartOrdinalも既に公開されていないが、これもctree()で解析できる。目的変数が順序カテゴリカルデータになっていれば特にオプションの指定は必要ない様だ。
データセットはパッケージRSADBEに同様の物が入っているのでこれを利用する。
library(RSADBE) data("lowbwt") lowbwt$Category <- factor(ifelse(lowbwt$BWT <= 2500, 3, ifelse(lowbwt$BWT <= 3000, 2, ifelse(lowbwt$BWT <= 3500, 1, 0))), ordered = TRUE) ord.rpart <- ctree(Category ~ AGE + LWT + RACE + SMOKE + PTL + HT + UI + FTV, data = lowbwt) ord.rpart # Model formula: # Category ~ AGE + LWT + RACE + SMOKE + PTL + HT + UI + FTV # # Fitted party: # [1] root # | [2] LWT <= 109: 3 (n = 42, err = 50.0%) # | [3] LWT > 109 # | | [4] UI <= 0 # | | | [5] SMOKE <= 0 # | | | | [6] RACE <= 1: 0 (n = 39, err = 43.6%) # | | | | [7] RACE > 1: 1 (n = 44, err = 63.6%) # | | | [8] SMOKE > 0: 3 (n = 47, err = 66.0%) # | | [9] UI > 0: 3 (n = 17, err = 41.2%) # # Number of inner nodes: 4 # Number of terminal nodes: 5 plot(ord.rpart)