ポアソン分布
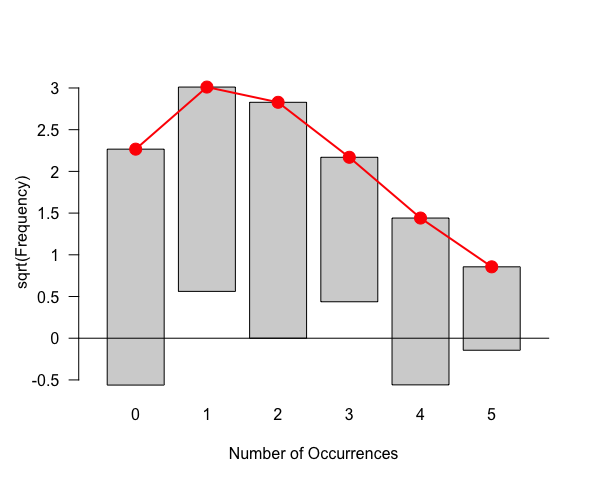
適合度検定はvcdパッケージのgoodfit()関数で行う。
> t <- 0:5 > x <- c(8, 6, 8, 3, 4, 1) > x <- matrix(c(x, t), nr = 6) > library(vcd) > result <- goodfit(x, type = "poisson", method = "MinChisq") > result Observed and fitted values for poisson distribution with parameters estimated by `MinChisq' count observed fitted pearson residual 0 8 5.1375937 1.262848928 1 6 9.0658624 -1.018235178 2 8 7.9988673 0.000400486 3 3 4.7049673 -0.786027501 4 4 2.0756111 1.335733618 5 1 0.7325299 -0.016953894
結果をplot()に渡すことで、結果を視覚的に判断することもできる。
ポアソン回帰分析
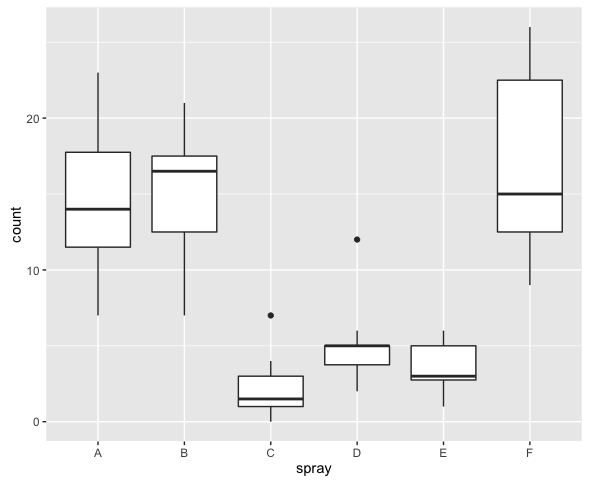
データセットInsectSpraysは殺虫剤ごとの死虫数*1をまとめたものである。このデータは平均値が大きいほど分散が大きく、ポアソン分布の特徴を持っているように見える。
ggplot(InsectSprays, aes(x = spray, y = count)) + geom_boxplot()
glm()による解析を行ってみる。
> result <- glm(count ~ spray, data = InsectSprays, family = "poisson") > summary(result) Call: glm(formula = count ~ spray, family = "poisson", data = InsectSprays) Deviance Residuals: Min 1Q Median 3Q Max -2.3852 -0.8876 -0.1482 0.6063 2.6922 Coefficients: Estimate Std. Error z value Pr(>|z|) (Intercept) 2.67415 0.07581 35.274 < 2e-16 *** sprayB 0.05588 0.10574 0.528 0.597 sprayC -1.94018 0.21389 -9.071 < 2e-16 *** sprayD -1.08152 0.15065 -7.179 7.03e-13 *** sprayE -1.42139 0.17192 -8.268 < 2e-16 *** sprayF 0.13926 0.10367 1.343 0.179 --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 (Dispersion parameter for poisson family taken to be 1) Null deviance: 409.041 on 71 degrees of freedom Residual deviance: 98.329 on 66 degrees of freedom AIC: 376.59 Number of Fisher Scoring iterations: 5
この結果から推定値を得るためには、例えばIntercept(=sprayA)であれば、sprayBであれば
というようにやるのだが、まとめて求めるにはpredict()関数を使用する。
> pred <- predict(result, newdata = data.frame(spray = LETTERS[1:6]), + type = "response") > pred 1 2 3 4 5 6 14.500000 15.333333 2.083333 4.916667 3.500000 16.666667
newdataには説明変数と同じ名前を持つ列に、予測したい説明変数の値をセットしたデータフレームを指定する。省略すると元のデータフレームの説明変数の値全てに対応する予測値が表示される。typeに"response"を指定することで、実際の予測値(上でexp()を使って計算した後の値)が表示される。指定しない場合は線形予測子の値(上でexp()に与えた中身)になる。
ついでに95%予測区間を求めてプロットしてみる。
> upr <- qpois(0.975, pred) > lwr <- qpois(0.025, pred) > ggplot(InsectSprays, aes(x = spray, y = count)) + geom_point() + + geom_pointrange(data = data.frame(spray = LETTERS[1:6], count = pred), + aes(ymin = lwr, ymax = upr), color = "blue")
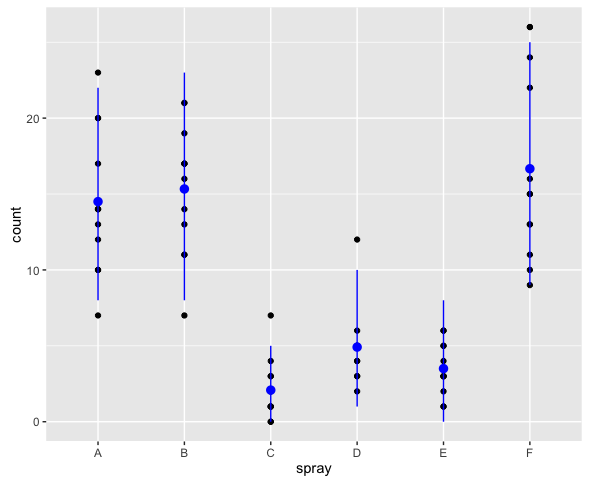
ちなみにこの予測区間はパラメータの標準誤差を考慮していないので、実際の予測区間よりたぶんちょっと狭い。そもそもこのような方法で予測区間を構成して良いものかどうかよく分からない。
オフセットによる調整法
まず、都道府県別の交通事故発生件数の一部を対象にポアソン回帰分析を行ってみる。
> accident <- c(45703, 8906, 7938, 12091, 7327, 9820, 11526, 6525) > pref <- c("A福岡", "B佐賀", "C長崎", "D熊本", + "E大分", "F宮崎", "G鹿児島", "H沖縄") > result1 <- glm(accident ~ pref, family = "poisson") > result1 Call: glm(formula = accident ~ pref, family = "poisson") Coefficients: (Intercept) prefB佐賀 prefC長崎 prefD熊本 prefE大分 10.730 -1.635 -1.751 -1.330 -1.831 prefF宮崎 prefG鹿児島 prefH沖縄 -1.538 -1.378 -1.947 Degrees of Freedom: 7 Total (i.e. Null); 0 Residual Null Deviance: 60920 Residual Deviance: 5.994e-12 AIC: 105.1
この結果を図示して解釈するためにまたpredict()を使ってみるが、typeに"terms"を指定すると、係数をスケールとした値が得られる。
具体的には"constant"属性に1つの値を持つベクトルが返ってくるが、constantの値とベクトルの各値を足したものが線形予測子である(result1のCoefficientsの値と比較してみると良い)。
> pred <- predict(result1, type = "terms") > pred pref 1 1.42600826 2 -0.20943048 3 -0.32449433 4 0.09630569 5 -0.40458953 6 -0.11173456 7 0.04844967 8 -0.52051473 attr(,"constant") [1] 9.303911
これをプロットしてみる。
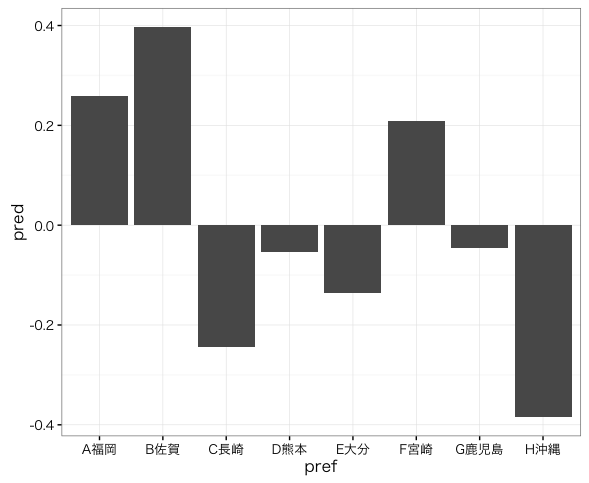
ggplot(data.frame(pref, pred), aes(x = pref, y = pred)) + geom_bar(stat = "identity") + theme_bw(base_family = "HiraKakuProN-W3") #Macで日本語を表示するために指定

圧倒的に福岡の係数が大きいが、これをもって福岡は交通事故が多いと判断して良いだろうか?
ここで、人口当たりの交通事故発生件数で解析することを試みる。
交通事故発生件数の平均を、人口を
とすると、次のようなモデルを解析することになる。
真数の商は対数の差であるので、右辺にを含めて解析することで
を解析することができる。
の様な項をオフセットと呼び、glmではoffset=に指定する。
> pop <- c(5056, 859, 1453, 1828, 1203, 1143, 1730, 1373) > result2 <- glm(accident ~ pref, offset = log(pop), family = poisson) > result2 Call: glm(formula = accident ~ pref, family = poisson, offset = log(pop)) Coefficients: (Intercept) prefB佐賀 prefC長崎 prefD熊本 prefE大分 2.20159 0.13712 -0.50356 -0.31235 -0.39484 prefF宮崎 prefG鹿児島 prefH沖縄 -0.05082 -0.30510 -0.64295 Degrees of Freedom: 7 Total (i.e. Null); 0 Residual Null Deviance: 5699 Residual Deviance: 8.338e-12 AIC: 105.1
先ほどと同様にプロットしてみると、結果は違ったものとなる。
> pred <- predict(result2, type = "terms") > ggplot(data.frame(pref, pred), + aes(x = pref, y = pred)) + + geom_bar(stat = "identity") + + theme_bw(base_family = "HiraKakuProN-W3")
過分散である場合の解析方法
ポアソン分布におけるパラメータが、観測のたびにガンマ分布に従って変動すると仮定する。
このような仮定にもとづいて乱数を発生させると、平均よりも分散が大きくなる。
> lambda <- rgamma(n, shape = 5, scale = 3) > y <- rpois(n, lambda) > mean(y) [1] 13.15 > var(y) [1] 49.08158
このような状況を過分散と呼ぶ。
次に、過分散する用量反応データを人工的に作成する。
n = 20 v = 3 dose <- 0:4 * 5 x <- rep(dose, n) a <- exp(3 + 0.05 * dose) / v lambda <- rgamma(5 * n, shape = a, scale = v) y <- rpois(5 * n, lambda) dataNB <- data.frame(x, y)
xで層別してプロットするとともに平均・分散を求めてみる。
> ggplot(dataNB, aes(x = x, y = y)) + geom_boxplot(aes(group = x)) > library(dplyr) > dataNB %>% group_by(x) %>% summarize(mean(y), var(y)) # A tibble: 5 x 3 x mean(y) var(y) <dbl> <dbl> <dbl> 1 0 18.60 43.09474 2 5 23.80 97.74737 3 10 30.70 56.22105 4 15 49.45 134.47105 5 20 52.70 212.64211
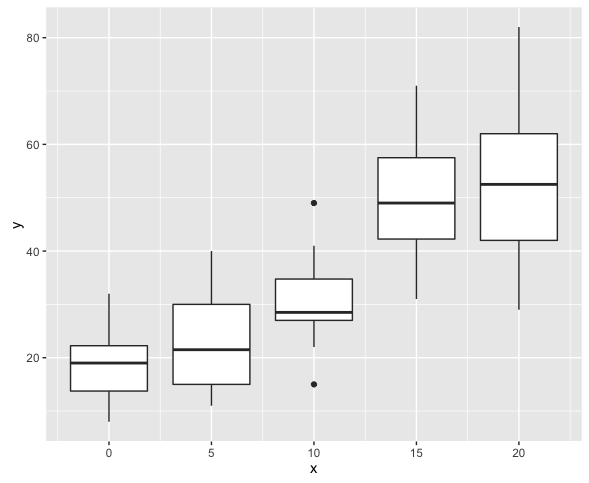
そのように作成したので当然ではあるが、過分散が確認できる。
このようなデータの解析には負の二項分布モデルを使用することができ、RではMASSパッケージのglm.nb()関数を用いる。使用方法はglm()とほとんど同じである。
> # 負の二項分布モデルによる分析 > result <- glm.nb(y ~ x, data = dataNB) > # ポアソン回帰分析 > result2 <- glm(y ~ x, data = dataNB, family = poisson) > result Call: glm.nb(formula = y ~ x, data = dataNB, init.theta = 15.79541194, link = log) Coefficients: (Intercept) x 2.91633 0.05636 Degrees of Freedom: 99 Total (i.e. Null); 98 Residual Null Deviance: 266.5 Residual Deviance: 102.7 AIC: 747.9 > result2 Call: glm(formula = y ~ x, family = poisson, data = dataNB) Coefficients: (Intercept) x 2.9285 0.0553 Degrees of Freedom: 99 Total (i.e. Null); 98 Residual Null Deviance: 825.1 Residual Deviance: 314.4 AIC: 845.6
ポアソン回帰分析に比べ、残差逸脱度、AIC共に小さくなっていることが分かる。
これもまたプロットしておこう。
# プロット ux <- seq(0, 20, by = 0.1) # 平均(mu)の計算 pred <- predict(result, newdata = data.frame(x = ux), type = "response") # 推定された形状パラメータ(theta)から予測区間を構成 # ※推定値の標準誤差を考慮していない upr <- qnbinom(0.975, size = result$theta, mu = pred) lwr <- qnbinom(0.025, size = result$theta, mu = pred) # plot ggplot(data.frame(ux, pred),aes(x = ux, y = pred)) + geom_line() + geom_ribbon(aes(ymin = lwr, ymax = upr), alpha = 0.25) + geom_point(data = dataNB, aes(x = x, y = y))
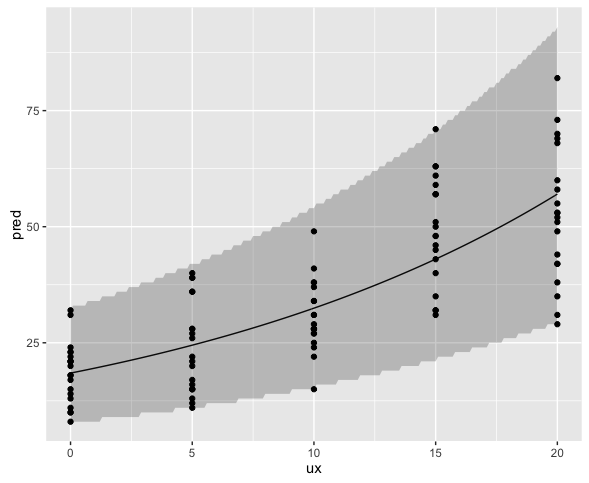
プロットの結果から見てもさほど問題は無いと思うが、予測区間はパラメータの標準誤差を考慮していないのできっと少し狭い。